Virulence genes of salmonella
Обновлено: 05.05.2024
Геном сальмонелл состоит из одной кольцевой хромосомы размером 4,8 млн пар нуклеотидов и ряда плазмид от 3 до 100 тысяч пар нуклеотидов (т.п.н.). Обуславливают генетическое разнообразие сальмонелл острова патогенности. Остров патогенности-1 (SPI-1) представляет собой участок ДНК размером 40 тысяч пар оснований, SPI-2 - 40 т.п.н. SPI-3 имеет размер 36 тысяч пар оснований, участвует в процессе внутриклеточного выживания и кодирует транспорт магния. SPI - 4 представляет собой 24 т.п.н. и участвует в адгезии к эпителиальным клеткам. SPI - 5 содержит менее 8 т.п.н. и необходим для инвазирования эпителия кишечника. SPI - 6 кодирует работу T6SS. SPI - 7 самый большой остров патогенности на сегодняшний день, содержит гены биосинтеза капсульного антигена Vi, отвечающего за вирулентность бактерии. SPI - 8 представляет собой фрагмент ДНК и является частью SPI-13. SPI-9 представляет собой локус размером 16 т.п.н. и содержит три гена, кодирующих T1SS. SPI-10 в S. typhi, состоит из 33 т.п.н. и включает несколько функционально несвязанных генов. SPI-11 участвует в интрамакрофагальной выживаемости сальмонелл. SPI – 12 кодирует специфические О-антигены. SPI - 13 состоит из 25 т.п.н. SPI-14 соответствует 9 т.п.н., кодирует цитоплазматические белки. SPI-15 состоит из 6,5 т.п.н., SPI - 16 из 4,5 т.п.н. SPI-17 кодирует остров в 5 т.п.н. SPI-18 размером 2,3 т.п.н., отвечает за инвазию сальмонелл в эпителиальные клетки кишечника человека. Другие острова патогенности не были идентифицированы как модули SPI, но они кодируют гены, ответственные за вирулентность бактерии.

1. Зайнуллин Л.И. Электрофосфоретические и антигенные свойства полипептидов сальмонелл и идентификация их геномов ПЦР : дис…канд. биол. наук Казань, 2003. – 157 с.
3. Колычев Н.М., Госманов Р.Г. Ветеринарная микробиология и иммунология. – 3-е изд., перераб. и доп. – М.: КолосС, 2003. – 432 с.
5. Abd E.l., Ghany M., Jansen A., Clare S., Hall L., Pickard D., Kingsley R., Dougan G. Candidate live, attenuated Salmonella entericaserotype Typhimurium vaccines with reduced fecal shedding are immunogenic and effective oral vaccines. // Infect Immun. 2007. Vol. 75. P. 1835–1842.
6. Bishop A., Baker S., Jenks S. et al. Analysis of the hypervariable region of the Salmonella enterica genome associated with tRNA(leuX). // J Bacteriol. 2005. Vol. 187. P. 2469–2482.
7. Blanc-Potard A., Groisman E. The Salmonella selC locus contains a pathogenicity island mediating intramacrophage survival. // EMBO J. 1997. Vol. 16. P. 5376–5385.
8. Buisan M., Rodriguez-Pena J.M., Rotger R. Restriction map of Salmonella enteritidis virulence plasmid and its homology with the plasmid of the Salmonella typhimurium // Microb Pathog. 1994. Vol. 16. P. 165 – 169.
9. Bukholm G., Figenschau K.J. Invasiveness of enterobacteria related to the presence of high molecular weight plasmids // Acta Pathol Microbiol Immunol Scand. 1988. Vol. 96. P. 30 – 36.
10. Chiu C., Tang P., Chu C. et al. The genome sequence of Salmonella enterica serovar Choleraesuis, a highly invasive and resistant zoonotic pathogen. // Nucleic Acids Res. 2005. Vol. 33. P. 1690–1698.
11. Chu C., Hong S.F., Tsai C., Lin W.S., Liu T.P., Ou J.T. Comparative physical and genetic maps of the virulence plasmids of Salmonella enterica serovars typhimurium, enteritidis, choleraesuis, and Dublin // Infect. Immun. 1999. Vol. P. 175 – 188.
13. Edwards R., Matlock B., Heffernan B., Maloy S. Genomic analysis and growth-phase-dependent regulation of the SEF14 fimbriae of Salmonella enterica serovar Enteritidis. // Microbiology. 2001. Vol. 147. P. 2705–2715.
14. Eriksson S., Lucchini S., Thompson A., Rhen M., Hinton J.C. Unravelling the biology of macrophage infection by gene expression profiling of intracellular Salmonella enterica. // Mol Microbiol. 2003. Vol. 47. P. 103–118.
15. Folkesson A., Advani A., Sukupolvi S., Pfeifer J., Normark S., Löfdahl S. Multiple insertions of fimbrial operons correlate with the evolution of Salmonella serovars responsible for human disease. // Mol Microbiol. 1999. Vol. 33. P. 612–622.
16. Galán J., Curtiss R. III Cloning and molecular characterization of genes whose products allow Salmonella typhimurium to penetrate tissue culture cells. // P Natl Acad Sci USA. 1989. Vol. 86. P. 6383–6387.
17. Gerlach R.G., Jackel D., Stecher B., Wagner C., Lupas A., Hardt W.D., Hensel M. Salmonella pathogenicity island 4 encodes a giant non-fimbrial adhesin and the cognate type 1 secretion system. // Cell Microbiol. 2007. Vol. 9. P. 1834–1850.
19. Gulig P.A., Doyle T.J. The Salmonella typhimurium virulence plasmid increases the growth rate of salmonellae in mice. // Infect Immun. 1993. Vol. 61. P. 504–511.
20. Gunn J., Alpuche-Aranda C., Loomis W., Belden W., Miller S. (1995) Characterization of the Salmonella typhimurium pagC/pagDchromosomal region. // J Bacteriol. 1995. Vol. 177. P. 5040–5047.
21. Haneda T., Ishii Y., Danbara H., Okada N. Genome-wide identification of novel genomic islands that contribute to Salmonellavirulence in mouse systemic infection. // FEMS Microbiol Lett. 2009. Vol. 297. P. 241–249.
22. Hansen-Wester I., Hensel M. Genome-based identification of chromosomal regions specific for Salmonella spp. // Infect Immun. 2002. Vol. 70. P. 2351–2360.
23. Hashimoto Y., Li N., Yokoyama H., Ezaki T. Complete nucleotide sequence and molecular characterization of ViaB region encoding Vi antigen in Salmonella typhi. // J Bacteriol. 1993. Vol. 175. P. 4456–4465.
24. Horiuchi S., Goto N., Ingki Y., Nakaya R. The 106-kilobase plasmid of Salmonella braenderup and the 100-kilobase plasmid Salmonella typhimurium are not necessary for the pathogenicity in experimental models // Microbiol Immunol. 1991. Vol. 35. P. 187 – 198.
25. Jones G., Rabert D., Svinarich D., Whitfield H. Association of adhesive, invasive, and virulent phenotypes of Salmonella typhimurium with autonomous 60-megadalton plasmids. // Infect Immun. 1982. Vol. 38. P. 476–486.
26. Kingsley R., Bäumler A. Pathogenicity islands and host adaptation of Salmonella serovars. // Curr Top Microbiol. 2002. Vol. 264. P. 67–87.
27. Kingsley R., Humphries A., Weening E. et al. Molecular and phenotypic analysis of the CS54 island of Salmonella entericaserotype Typhimurium: identification of intestinal colonization and persistence determinants. // Infect Immun. 2003. Vol. 71. P. 629–640.
28. Kotetishvili M., Stine O.C., Kreger A., Morris J.G., Sulakvedze A. Multilocus Sequence Typing for Characterization of Clinical and Environmental Salmonella Strains // J Clin Microbiol. 2002. Vol. 40. P. 1626 – 1635.
29. Lesnick M., Reiner N., Fierer J., Guiney D. The Salmonella spvB virulence gene encodes an enzyme that ADP-ribosylates actin and destabilizes the cytoskeleton of eukaryotic cells. // Mol Microbiol. 2001. Vol. 39. P. 1464–1470.
30. McClelland M., Sanderson K., Spieth J. et al. Complete genome sequence of Salmonella enterica serovar Typhimurium LT2. // Nature. 2001. Vol. 413. P. 852–856.
31. Miao E., Scherer C., Tsolis R., Kingsley R., Adams L., Bäumler A., Miller S. Salmonella typhimurium leucine-rich repeat proteins are targeted to the SPI1 and SPI2 type III secretion systems. // Mol Microbiol. 1999. Vol. 34. P. 850–864.
32. Miller S.I., Kukral A.M., Mekalanos J.J. A two-component regulatory system (phoP phoQ) controls Salmonella typhimuriumvirulence. // P Natl Acad Sci USA. 1989. Vol. 86. P. 5054–5058.
33. Morgan E. Salmonella pathogenicity islands. Salmonella Molecular Biology and Pathogenesis (Rhen M., Maskell D., Mastroeni P., Threlfall J. et al.) // Horizon Bioscience, Norfolk. 2007. Рp. 67–88.
34. Morgan E., Campbell J.D., Rowe S.C. et al. Identification of host-specific colonization factors of Salmonella enterica serovar Typhimurium. // Mol Microbiol. 2004. Vol. 54. P. 994–1010.
35. Parkhill J., Dougan G., James K. et al. Complete genome sequence of a multiple drug resistant Salmonella enterica serovar Typhi CT18. // Nature. 2001. Vol. 413. P. 848–852.
36. Pickard D., Wain J., Baker S. et al. Composition, acquisition, and distribution of the Vi exopolysaccharide-encoding Salmonella enterica pathogenicity island SPI-7. // J Bacteriol. 2003. Vol. 185. P. 5055–5065.
37. Shah D., Lee M., Park J., Lee J., Eo S., Kwon J., Chae J. Identification of Salmonella gallinarum virulence genes in a chicken infection model using PCR-based signature-tagged mutagenesis. // Microbiology. 2005. Vol. 151. P. 3957–3968.
38. Shi L., Adkins J.N., Coleman J.R. et al. Proteomic analysis of Salmonella enterica serovar typhimurium isolated from RAW 264.7 macrophages: identification of a novel protein that contributes to the replication of serovar typhimurium inside macrophages.// J Biol Chem. 2006. Vol. 281. P. 29131–29140.
39. Slauch J., Lee A., Mahan M., Mekalanos J. Molecular characterization of the oafA locus responsible for acetylation of Salmonella typhimurium O-antigen: oafA is a member of a family of integral membrane trans-acylases. // J Bacteriol. 1996. Vol. 178. P. 5904–5909.
40. Taira S. and Rhen M. Molecular organization of genes constituting the virulence determinant on the Salmonella typhimurium 96 kilobase pair plasmid // FEBS Letters. 1989. Vol. 257. P. 274 – 278.
41. Townsend S., Kramer N., Edwards R. et al. Salmonella enterica serovar Typhi possesses a unique repertoire of fimbrial gene sequences. // Infect Immun. 2001. Vol. 69. P. 2894–2901.
42. Vernikos G., Parkhill J. Interpolated variable order motifs for identification of horizontally acquired DNA: revisiting the Salmonella pathogenicity islands. // Bioinformatics. 2006. Vol. 22. P. 2196–2203.
43. Williamson C.M., Baird G.D., Manning E.J. A common virulence region on plasmids from eleven serotyps of Salmonella // J Gen Microbiol. 1988. Vol. 134. P. 975 – 982.
44. Wong K., McClelland M., Stillwell L., Sisk E., Thurston S., Saffer J. Identification and sequence analysis of a 27-kilobase chromosomal fragment containing a Salmonella pathogenicity island located at 92 minutes on the chromosome map of Salmonella enterica serovar typhimurium LT2. // Infect Immun. 1998. Vol. 66. P. 3365–3371.
45. Wood M., Jones M., Watson P., Hedges S., Wallis T., Galyov E. Identification of a pathogenicity island required for Salmonellaenteropathogenicity. // Mol Microbiol. 1998. Vol. 29. P. 883–891.
46. Woodward M.J., McLaren I., Wray C. Distribution of virulence plasmids within Salmonella // J Gen Microbiol. 1989. Vol. 135. P. 503 – 511.
47. Zhang X.L., Tsui I.S., Yip C.M. et al. Salmonella enterica serovar Typhi uses type IVB pili to enter human intestinal epithelial cells. // Infect Immun. 2000. Vol. 68. P. 3067–3073.
Сальмонеллы - лактозонегативные грамотрицательные палочки удлиненной формы, с закругленными концами длиной 1. 4 и шириной 0,3. 0,8 мкм. Подавляющее большинство представителей рода Salmonella подвижны (за исключением S. gallinarum-рullorum), органами их движения являются 4-5 жгутиков, расположенных равномерно по всей поверхности микробной клетки [1, 3, 12]. Геном сальмонелл состоит из одной кольцевой хромосомы размером 4,8 млн пар нуклеотидов и ряда плазмид от 3 до 100 тысяч пар нуклеотидов (т.п.н.), при этом обнаружено значительное сходство между плазмидами вирулентности различных серотипов [8, 43, 46].
Геном серовара S.typhimurium полностью расшифрован в 2001 г.[4, 31, 42]. Ранее было установлено, что большинство штаммов S.typhimurium состоят из вирулентных плазмид размером около 90 т.п.н. [18, 19, 25].
Плазмида S. dublin размером 76 т.п.н., включает 3 района вирулентности [43]. По данным азиатских ученых плазмида S. choleraesuis варьирует от 50 до 110 т.п.н., а у S. enteritidis - 60 т.п.н. [11]. Известно, что некоторые штаммы сальмонелл не имеют плазмид вирулентности, при этом они не теряют своей инвазивной способности [9, 24]. Хромосома S. enterica очень сходна с таковой E.coli и состоит из единственной циркулярной молекулы ДНК размером около 4 млн пар оснований. Среднее суммарное содержание в ней цитозина и гуанина составляет 52% [4].
Информацию о ферментах, ответственных за синтез и сборку полисахаридных частей О-антигена, кодирует кластер генов в локусе rfb хромосомы. Изменения О-антигена происходят в результате лизогенной конверсии хромосомных генов бактериофагами или мутаций [4, 39].
Образование жгутиков зависит от 3 классов генов, функции которых контролируются рядом регуляторов, в т.ч. сигма-фактором FliA и его антагонистом антисигма-фактором FlgM. У многих сероваров сальмонелл 2 набора генов флагеллина, из которых в экспрессии белка задействована только 1 аллель. Опероны, контролирующие синтез каждой фазы жгутикового антигена, также кодируют репрессор синтеза другой фазы флагеллина.
У S. enterica имеется 4 пильных оперона: fim (типа 1), lpf (длинных полярных пилей), pef (плазмидокодируемых пилей) и agf (тонких агрегативных пилей) [4].
Хромосомный локус inv включает 14 генов, наиболее известными из них являются invA, invE. Продукты данных генов необходимы для инвазии бактерий через эпителиальные клетки кишечника Часть этих генов гомологична генам E.coli, регулирующим сборку жгутиков [16, 28].
В тесной связи с локусом inv функционирует рядом лежащий локус Spa. Между его 12 генами и генами плазмиды вирулентности шигелл выявлена высокая степень идентичности и сходная последовательность локализации. Сходство ряда генов этих локусов с генами LcrD, LcrE и YscA йерсиний позволяет предположить, что транспортировка инвазивных протеинов сальмонелл осуществляется по тем же механизмам, что и экспорт жгутиковых белков.
Упомянутые локусы включают в себя острова патогенности, обуславливающие генетическое разнообразие сальмонелл. Сальмонеллы содержат ряд генов вирулентности, известных как модули или острова патогенности. На сегодняшний день известен двадцать один модуль SPI [35, 42]. Остров патогенности-1 (SPI-1) представляет собой участок ДНК размером 40 тысяч пар оснований [16]. Данный модуль кодирует 33 протеина, в т.ч. компоненты секреционной системы типа III (T3SS), регуляторные и секреционные эффекторные протеины, а также опероны. T3SS используются бактериями для введения белков, называемых эффекторами, непосредственно внутрь клеток-хозяев, которые будут выступать в качестве медиаторов вторжения клеток и модификаций, способствующих внутриклеточному росту [35]. Изменение генов invA, invF, invG, hilA, sipC, sipD, spaR и orgB этой системы ведет к 16-100-кратному снижению вирулентности S. enterica. Остров патогенности 2 (SPI-2) имеет размер 40 тысяч пар оснований. Он кодирует второй вид секреционной системы типа III, который участвует во внутриклеточной выживаемости и системе сборки жгутиков. Приобретение SPI-2 позволило сальмонеллам перейти от выживания к репродукции в клетках хозяина и от местной инфекции пищеварительного тракта к системной диссеминации. Остров патогенности-3 (SPI-3) имеет размер 36 тысяч пар оснований, участвует в процессе внутриклеточного выживания и кодирует транспорт магния. Только один его ген (mgtC) ассоциирован с вирулентностью - кодируемый им продукт обеспечивает рост бактерии в макрофагах и проявление системной вирулентности за счет адаптации к условиям низкого содержания ионов магния и низкому рН фагосомы [7]. SPI - 4 представляет собой 24 т.п.н. и участвует в адгезии к эпителиальным клеткам [17, 33, 34, 44]. SPI - 5 является небольшим островом патогенности размером менее 8 т.п.н., он необходим для инвазирования эпителия кишечника [44].SPI - 6 кодирует работу T6SS, safABCD фимбриальный кластер генов и инвазивный pagN [15, 41]. SPI - 7 самый большой остров патогенности на сегодняшний день (отсутствует в S . Typhimurium, но присутствует в S .Typhi) [35]. В S . Typhi размер данного модуля составляет 134 т.п.н., что соответствует примерно 150 генов [22, 36]. Этот остров содержит гены биосинтеза капсульного антигена Vi, отвечающего за вирулентность бактерии [23, 47]. SPI - 8 представляет собой фрагмент ДНК и является частью SPI-13 [35]. SPI-9 представляет собой локус размером 16 т.п.н. и содержит три гена, кодирующих T1SS [33, 34]. SPI-10 наиболее полно изучен в S. typhi, состоит из 33 т.п.н. и включает несколько функционально несвязанных генов [6, 13, 35]. Опыты Haneda et al.(2009) показали, что удаление SPI-10 из S. typhimurium штамм 14028 приводит к ослаблению вирулентности сальмонелл [21]. SPI-11 был первоначально идентифицирован в геномной последовательности серовара S. choleraesuis, его размер соответствовал 14 т.п.н. Несколько короче данный остров патогенности в S. typhimurium (6,7 т.п.н.) и в S. typhi (10 т.п.н.). SPI-11 участвует в интрамакрофагальной выживаемости сальмонелл [10, 20, 32]. SPI - 12 состоит из 15,8 т.п.н. в S. typhimurium и 6,3 т.п.н. в S. typhi, кодирует специфические О-антигены [22, 31, 39]. SPI - 13 был первоначально идентифицирован в серотипе S. gallinarum. Состоит из 25 т.п.н., однако 8 т.п.н. несут различную функциональную нагрузку в разных серотипах сальмонелл. Отвечают за гены, кодирующие работу лиазы, гидролазы, оксидазы; вирулентность бактерии; репликацию внутри макрофагов [21, 37, 38]. SPI-14 соответствует 9 т.п.н., (отсутствует в S. Typhi) [33, 37]. Функция SPI-14 на сегодняшний день невыяснена, но известно, что данный остров патогенности кодирует цитоплазматические белки [14]. SPI-15 остров патогенности размером 6,5 т.п.н. (отсутствует в S. typhimurium). SPI - 16 находится в S. typhimurium и S. typhi, размер его составляет 4,5 т.п.н. SPI-17 кодирует остров в 5 т.п.н. (отсутствует в S. typhimurium) [42]. SPI-18 был идентифицирован в S. Typhi, размером 2,3 т.п.н., в опытах in vitro установлено, что модуль отвечает за инвазию сальмонелл в эпителиальные клетки кишечника человека. Другие острова патогенности не были идентифицированы как модули SPI, но они кодируют гены, ответственные за вирулентность бактерии. [5, 26, 27].
Регуляторная система PhoP/PhoQ регулирует изменения липополисахаридов самой бактерии, что повышает ее резистентность к меняющимся условиям внешней среды и антимикробным препаратам, а также ведет к затруднению распознавания липополисахарида иммунной системой.
Гены spv (spvR, spvA, spvB, spvC и spvD) у S.typhimurium и S.enteritidis локализуются в крупной (размером 50-100 тысяч пар оснований) плазмиде, а у остальных сероваров в хромосоме. Кодируемые ими факторы обеспечивают распространение сальмонелл по организму, репродукцию в моноцитах, а также индукцию апоптоза последних.
Ген shdA размером в 6105 пар оснований проявляет гомологию с участками ДНК шигелл и диареегенных штаммов E.coli. Он обеспечил S. enterica адаптацию к теплокровным животным и регулирование интенсивности выделения бактерии с фекалиями.
Уникальный ген sifA состоит из 300 пар оснований и имеет более низкое суммарное содержание гуанина и цитозина (41%), чем другие части ДНК. Отвечает за образование филаментов, связывающих агента с мембраной фагосомы клеток эукариотов [4].
Итак, патогенные свойства микроорганизмов детерминированы в геноме. Причем некоторые из них имеют одну группу детерминант, другие - четыре, чем и определяются различия в патогенности сальмонелл [2].
Рецензенты:
Геном сальмонелл состоит из одной кольцевой хромосомы размером 4,8 млн пар нуклеотидов и ряда плазмид от 3 до 100 тысяч пар нуклеотидов (т.п.н.). Обуславливают генетическое разнообразие сальмонелл острова патогенности. Остров патогенности-1 (SPI-1) представляет собой участок ДНК размером 40 тысяч пар оснований, SPI-2 - 40 т.п.н. SPI-3 имеет размер 36 тысяч пар оснований, участвует в процессе внутриклеточного выживания и кодирует транспорт магния. SPI - 4 представляет собой 24 т.п.н. и участвует в адгезии к эпителиальным клеткам. SPI - 5 содержит менее 8 т.п.н. и необходим для инвазирования эпителия кишечника. SPI - 6 кодирует работу T6SS. SPI - 7 самый большой остров патогенности на сегодняшний день, содержит гены биосинтеза капсульного антигена Vi, отвечающего за вирулентность бактерии. SPI - 8 представляет собой фрагмент ДНК и является частью SPI-13. SPI-9 представляет собой локус размером 16 т.п.н. и содержит три гена, кодирующих T1SS. SPI-10 в S. typhi, состоит из 33 т.п.н. и включает несколько функционально несвязанных генов. SPI-11 участвует в интрамакрофагальной выживаемости сальмонелл. SPI – 12 кодирует специфические О-антигены. SPI - 13 состоит из 25 т.п.н. SPI-14 соответствует 9 т.п.н., кодирует цитоплазматические белки. SPI-15 состоит из 6,5 т.п.н., SPI - 16 из 4,5 т.п.н. SPI-17 кодирует остров в 5 т.п.н. SPI-18 размером 2,3 т.п.н., отвечает за инвазию сальмонелл в эпителиальные клетки кишечника человека. Другие острова патогенности не были идентифицированы как модули SPI, но они кодируют гены, ответственные за вирулентность бактерии.

1. Зайнуллин Л.И. Электрофосфоретические и антигенные свойства полипептидов сальмонелл и идентификация их геномов ПЦР : дис…канд. биол. наук Казань, 2003. – 157 с.
3. Колычев Н.М., Госманов Р.Г. Ветеринарная микробиология и иммунология. – 3-е изд., перераб. и доп. – М.: КолосС, 2003. – 432 с.
5. Abd E.l., Ghany M., Jansen A., Clare S., Hall L., Pickard D., Kingsley R., Dougan G. Candidate live, attenuated Salmonella entericaserotype Typhimurium vaccines with reduced fecal shedding are immunogenic and effective oral vaccines. // Infect Immun. 2007. Vol. 75. P. 1835–1842.
6. Bishop A., Baker S., Jenks S. et al. Analysis of the hypervariable region of the Salmonella enterica genome associated with tRNA(leuX). // J Bacteriol. 2005. Vol. 187. P. 2469–2482.
7. Blanc-Potard A., Groisman E. The Salmonella selC locus contains a pathogenicity island mediating intramacrophage survival. // EMBO J. 1997. Vol. 16. P. 5376–5385.
8. Buisan M., Rodriguez-Pena J.M., Rotger R. Restriction map of Salmonella enteritidis virulence plasmid and its homology with the plasmid of the Salmonella typhimurium // Microb Pathog. 1994. Vol. 16. P. 165 – 169.
9. Bukholm G., Figenschau K.J. Invasiveness of enterobacteria related to the presence of high molecular weight plasmids // Acta Pathol Microbiol Immunol Scand. 1988. Vol. 96. P. 30 – 36.
10. Chiu C., Tang P., Chu C. et al. The genome sequence of Salmonella enterica serovar Choleraesuis, a highly invasive and resistant zoonotic pathogen. // Nucleic Acids Res. 2005. Vol. 33. P. 1690–1698.
11. Chu C., Hong S.F., Tsai C., Lin W.S., Liu T.P., Ou J.T. Comparative physical and genetic maps of the virulence plasmids of Salmonella enterica serovars typhimurium, enteritidis, choleraesuis, and Dublin // Infect. Immun. 1999. Vol. P. 175 – 188.
13. Edwards R., Matlock B., Heffernan B., Maloy S. Genomic analysis and growth-phase-dependent regulation of the SEF14 fimbriae of Salmonella enterica serovar Enteritidis. // Microbiology. 2001. Vol. 147. P. 2705–2715.
14. Eriksson S., Lucchini S., Thompson A., Rhen M., Hinton J.C. Unravelling the biology of macrophage infection by gene expression profiling of intracellular Salmonella enterica. // Mol Microbiol. 2003. Vol. 47. P. 103–118.
15. Folkesson A., Advani A., Sukupolvi S., Pfeifer J., Normark S., Löfdahl S. Multiple insertions of fimbrial operons correlate with the evolution of Salmonella serovars responsible for human disease. // Mol Microbiol. 1999. Vol. 33. P. 612–622.
16. Galán J., Curtiss R. III Cloning and molecular characterization of genes whose products allow Salmonella typhimurium to penetrate tissue culture cells. // P Natl Acad Sci USA. 1989. Vol. 86. P. 6383–6387.
17. Gerlach R.G., Jackel D., Stecher B., Wagner C., Lupas A., Hardt W.D., Hensel M. Salmonella pathogenicity island 4 encodes a giant non-fimbrial adhesin and the cognate type 1 secretion system. // Cell Microbiol. 2007. Vol. 9. P. 1834–1850.
19. Gulig P.A., Doyle T.J. The Salmonella typhimurium virulence plasmid increases the growth rate of salmonellae in mice. // Infect Immun. 1993. Vol. 61. P. 504–511.
20. Gunn J., Alpuche-Aranda C., Loomis W., Belden W., Miller S. (1995) Characterization of the Salmonella typhimurium pagC/pagDchromosomal region. // J Bacteriol. 1995. Vol. 177. P. 5040–5047.
21. Haneda T., Ishii Y., Danbara H., Okada N. Genome-wide identification of novel genomic islands that contribute to Salmonellavirulence in mouse systemic infection. // FEMS Microbiol Lett. 2009. Vol. 297. P. 241–249.
22. Hansen-Wester I., Hensel M. Genome-based identification of chromosomal regions specific for Salmonella spp. // Infect Immun. 2002. Vol. 70. P. 2351–2360.
23. Hashimoto Y., Li N., Yokoyama H., Ezaki T. Complete nucleotide sequence and molecular characterization of ViaB region encoding Vi antigen in Salmonella typhi. // J Bacteriol. 1993. Vol. 175. P. 4456–4465.
24. Horiuchi S., Goto N., Ingki Y., Nakaya R. The 106-kilobase plasmid of Salmonella braenderup and the 100-kilobase plasmid Salmonella typhimurium are not necessary for the pathogenicity in experimental models // Microbiol Immunol. 1991. Vol. 35. P. 187 – 198.
25. Jones G., Rabert D., Svinarich D., Whitfield H. Association of adhesive, invasive, and virulent phenotypes of Salmonella typhimurium with autonomous 60-megadalton plasmids. // Infect Immun. 1982. Vol. 38. P. 476–486.
26. Kingsley R., Bäumler A. Pathogenicity islands and host adaptation of Salmonella serovars. // Curr Top Microbiol. 2002. Vol. 264. P. 67–87.
27. Kingsley R., Humphries A., Weening E. et al. Molecular and phenotypic analysis of the CS54 island of Salmonella entericaserotype Typhimurium: identification of intestinal colonization and persistence determinants. // Infect Immun. 2003. Vol. 71. P. 629–640.
28. Kotetishvili M., Stine O.C., Kreger A., Morris J.G., Sulakvedze A. Multilocus Sequence Typing for Characterization of Clinical and Environmental Salmonella Strains // J Clin Microbiol. 2002. Vol. 40. P. 1626 – 1635.
29. Lesnick M., Reiner N., Fierer J., Guiney D. The Salmonella spvB virulence gene encodes an enzyme that ADP-ribosylates actin and destabilizes the cytoskeleton of eukaryotic cells. // Mol Microbiol. 2001. Vol. 39. P. 1464–1470.
30. McClelland M., Sanderson K., Spieth J. et al. Complete genome sequence of Salmonella enterica serovar Typhimurium LT2. // Nature. 2001. Vol. 413. P. 852–856.
31. Miao E., Scherer C., Tsolis R., Kingsley R., Adams L., Bäumler A., Miller S. Salmonella typhimurium leucine-rich repeat proteins are targeted to the SPI1 and SPI2 type III secretion systems. // Mol Microbiol. 1999. Vol. 34. P. 850–864.
32. Miller S.I., Kukral A.M., Mekalanos J.J. A two-component regulatory system (phoP phoQ) controls Salmonella typhimuriumvirulence. // P Natl Acad Sci USA. 1989. Vol. 86. P. 5054–5058.
33. Morgan E. Salmonella pathogenicity islands. Salmonella Molecular Biology and Pathogenesis (Rhen M., Maskell D., Mastroeni P., Threlfall J. et al.) // Horizon Bioscience, Norfolk. 2007. Рp. 67–88.
34. Morgan E., Campbell J.D., Rowe S.C. et al. Identification of host-specific colonization factors of Salmonella enterica serovar Typhimurium. // Mol Microbiol. 2004. Vol. 54. P. 994–1010.
35. Parkhill J., Dougan G., James K. et al. Complete genome sequence of a multiple drug resistant Salmonella enterica serovar Typhi CT18. // Nature. 2001. Vol. 413. P. 848–852.
36. Pickard D., Wain J., Baker S. et al. Composition, acquisition, and distribution of the Vi exopolysaccharide-encoding Salmonella enterica pathogenicity island SPI-7. // J Bacteriol. 2003. Vol. 185. P. 5055–5065.
37. Shah D., Lee M., Park J., Lee J., Eo S., Kwon J., Chae J. Identification of Salmonella gallinarum virulence genes in a chicken infection model using PCR-based signature-tagged mutagenesis. // Microbiology. 2005. Vol. 151. P. 3957–3968.
38. Shi L., Adkins J.N., Coleman J.R. et al. Proteomic analysis of Salmonella enterica serovar typhimurium isolated from RAW 264.7 macrophages: identification of a novel protein that contributes to the replication of serovar typhimurium inside macrophages.// J Biol Chem. 2006. Vol. 281. P. 29131–29140.
39. Slauch J., Lee A., Mahan M., Mekalanos J. Molecular characterization of the oafA locus responsible for acetylation of Salmonella typhimurium O-antigen: oafA is a member of a family of integral membrane trans-acylases. // J Bacteriol. 1996. Vol. 178. P. 5904–5909.
40. Taira S. and Rhen M. Molecular organization of genes constituting the virulence determinant on the Salmonella typhimurium 96 kilobase pair plasmid // FEBS Letters. 1989. Vol. 257. P. 274 – 278.
41. Townsend S., Kramer N., Edwards R. et al. Salmonella enterica serovar Typhi possesses a unique repertoire of fimbrial gene sequences. // Infect Immun. 2001. Vol. 69. P. 2894–2901.
42. Vernikos G., Parkhill J. Interpolated variable order motifs for identification of horizontally acquired DNA: revisiting the Salmonella pathogenicity islands. // Bioinformatics. 2006. Vol. 22. P. 2196–2203.
43. Williamson C.M., Baird G.D., Manning E.J. A common virulence region on plasmids from eleven serotyps of Salmonella // J Gen Microbiol. 1988. Vol. 134. P. 975 – 982.
44. Wong K., McClelland M., Stillwell L., Sisk E., Thurston S., Saffer J. Identification and sequence analysis of a 27-kilobase chromosomal fragment containing a Salmonella pathogenicity island located at 92 minutes on the chromosome map of Salmonella enterica serovar typhimurium LT2. // Infect Immun. 1998. Vol. 66. P. 3365–3371.
45. Wood M., Jones M., Watson P., Hedges S., Wallis T., Galyov E. Identification of a pathogenicity island required for Salmonellaenteropathogenicity. // Mol Microbiol. 1998. Vol. 29. P. 883–891.
46. Woodward M.J., McLaren I., Wray C. Distribution of virulence plasmids within Salmonella // J Gen Microbiol. 1989. Vol. 135. P. 503 – 511.
47. Zhang X.L., Tsui I.S., Yip C.M. et al. Salmonella enterica serovar Typhi uses type IVB pili to enter human intestinal epithelial cells. // Infect Immun. 2000. Vol. 68. P. 3067–3073.
Сальмонеллы - лактозонегативные грамотрицательные палочки удлиненной формы, с закругленными концами длиной 1. 4 и шириной 0,3. 0,8 мкм. Подавляющее большинство представителей рода Salmonella подвижны (за исключением S. gallinarum-рullorum), органами их движения являются 4-5 жгутиков, расположенных равномерно по всей поверхности микробной клетки [1, 3, 12]. Геном сальмонелл состоит из одной кольцевой хромосомы размером 4,8 млн пар нуклеотидов и ряда плазмид от 3 до 100 тысяч пар нуклеотидов (т.п.н.), при этом обнаружено значительное сходство между плазмидами вирулентности различных серотипов [8, 43, 46].
Геном серовара S.typhimurium полностью расшифрован в 2001 г.[4, 31, 42]. Ранее было установлено, что большинство штаммов S.typhimurium состоят из вирулентных плазмид размером около 90 т.п.н. [18, 19, 25].
Плазмида S. dublin размером 76 т.п.н., включает 3 района вирулентности [43]. По данным азиатских ученых плазмида S. choleraesuis варьирует от 50 до 110 т.п.н., а у S. enteritidis - 60 т.п.н. [11]. Известно, что некоторые штаммы сальмонелл не имеют плазмид вирулентности, при этом они не теряют своей инвазивной способности [9, 24]. Хромосома S. enterica очень сходна с таковой E.coli и состоит из единственной циркулярной молекулы ДНК размером около 4 млн пар оснований. Среднее суммарное содержание в ней цитозина и гуанина составляет 52% [4].
Информацию о ферментах, ответственных за синтез и сборку полисахаридных частей О-антигена, кодирует кластер генов в локусе rfb хромосомы. Изменения О-антигена происходят в результате лизогенной конверсии хромосомных генов бактериофагами или мутаций [4, 39].
Образование жгутиков зависит от 3 классов генов, функции которых контролируются рядом регуляторов, в т.ч. сигма-фактором FliA и его антагонистом антисигма-фактором FlgM. У многих сероваров сальмонелл 2 набора генов флагеллина, из которых в экспрессии белка задействована только 1 аллель. Опероны, контролирующие синтез каждой фазы жгутикового антигена, также кодируют репрессор синтеза другой фазы флагеллина.
У S. enterica имеется 4 пильных оперона: fim (типа 1), lpf (длинных полярных пилей), pef (плазмидокодируемых пилей) и agf (тонких агрегативных пилей) [4].
Хромосомный локус inv включает 14 генов, наиболее известными из них являются invA, invE. Продукты данных генов необходимы для инвазии бактерий через эпителиальные клетки кишечника Часть этих генов гомологична генам E.coli, регулирующим сборку жгутиков [16, 28].
В тесной связи с локусом inv функционирует рядом лежащий локус Spa. Между его 12 генами и генами плазмиды вирулентности шигелл выявлена высокая степень идентичности и сходная последовательность локализации. Сходство ряда генов этих локусов с генами LcrD, LcrE и YscA йерсиний позволяет предположить, что транспортировка инвазивных протеинов сальмонелл осуществляется по тем же механизмам, что и экспорт жгутиковых белков.
Упомянутые локусы включают в себя острова патогенности, обуславливающие генетическое разнообразие сальмонелл. Сальмонеллы содержат ряд генов вирулентности, известных как модули или острова патогенности. На сегодняшний день известен двадцать один модуль SPI [35, 42]. Остров патогенности-1 (SPI-1) представляет собой участок ДНК размером 40 тысяч пар оснований [16]. Данный модуль кодирует 33 протеина, в т.ч. компоненты секреционной системы типа III (T3SS), регуляторные и секреционные эффекторные протеины, а также опероны. T3SS используются бактериями для введения белков, называемых эффекторами, непосредственно внутрь клеток-хозяев, которые будут выступать в качестве медиаторов вторжения клеток и модификаций, способствующих внутриклеточному росту [35]. Изменение генов invA, invF, invG, hilA, sipC, sipD, spaR и orgB этой системы ведет к 16-100-кратному снижению вирулентности S. enterica. Остров патогенности 2 (SPI-2) имеет размер 40 тысяч пар оснований. Он кодирует второй вид секреционной системы типа III, который участвует во внутриклеточной выживаемости и системе сборки жгутиков. Приобретение SPI-2 позволило сальмонеллам перейти от выживания к репродукции в клетках хозяина и от местной инфекции пищеварительного тракта к системной диссеминации. Остров патогенности-3 (SPI-3) имеет размер 36 тысяч пар оснований, участвует в процессе внутриклеточного выживания и кодирует транспорт магния. Только один его ген (mgtC) ассоциирован с вирулентностью - кодируемый им продукт обеспечивает рост бактерии в макрофагах и проявление системной вирулентности за счет адаптации к условиям низкого содержания ионов магния и низкому рН фагосомы [7]. SPI - 4 представляет собой 24 т.п.н. и участвует в адгезии к эпителиальным клеткам [17, 33, 34, 44]. SPI - 5 является небольшим островом патогенности размером менее 8 т.п.н., он необходим для инвазирования эпителия кишечника [44].SPI - 6 кодирует работу T6SS, safABCD фимбриальный кластер генов и инвазивный pagN [15, 41]. SPI - 7 самый большой остров патогенности на сегодняшний день (отсутствует в S . Typhimurium, но присутствует в S .Typhi) [35]. В S . Typhi размер данного модуля составляет 134 т.п.н., что соответствует примерно 150 генов [22, 36]. Этот остров содержит гены биосинтеза капсульного антигена Vi, отвечающего за вирулентность бактерии [23, 47]. SPI - 8 представляет собой фрагмент ДНК и является частью SPI-13 [35]. SPI-9 представляет собой локус размером 16 т.п.н. и содержит три гена, кодирующих T1SS [33, 34]. SPI-10 наиболее полно изучен в S. typhi, состоит из 33 т.п.н. и включает несколько функционально несвязанных генов [6, 13, 35]. Опыты Haneda et al.(2009) показали, что удаление SPI-10 из S. typhimurium штамм 14028 приводит к ослаблению вирулентности сальмонелл [21]. SPI-11 был первоначально идентифицирован в геномной последовательности серовара S. choleraesuis, его размер соответствовал 14 т.п.н. Несколько короче данный остров патогенности в S. typhimurium (6,7 т.п.н.) и в S. typhi (10 т.п.н.). SPI-11 участвует в интрамакрофагальной выживаемости сальмонелл [10, 20, 32]. SPI - 12 состоит из 15,8 т.п.н. в S. typhimurium и 6,3 т.п.н. в S. typhi, кодирует специфические О-антигены [22, 31, 39]. SPI - 13 был первоначально идентифицирован в серотипе S. gallinarum. Состоит из 25 т.п.н., однако 8 т.п.н. несут различную функциональную нагрузку в разных серотипах сальмонелл. Отвечают за гены, кодирующие работу лиазы, гидролазы, оксидазы; вирулентность бактерии; репликацию внутри макрофагов [21, 37, 38]. SPI-14 соответствует 9 т.п.н., (отсутствует в S. Typhi) [33, 37]. Функция SPI-14 на сегодняшний день невыяснена, но известно, что данный остров патогенности кодирует цитоплазматические белки [14]. SPI-15 остров патогенности размером 6,5 т.п.н. (отсутствует в S. typhimurium). SPI - 16 находится в S. typhimurium и S. typhi, размер его составляет 4,5 т.п.н. SPI-17 кодирует остров в 5 т.п.н. (отсутствует в S. typhimurium) [42]. SPI-18 был идентифицирован в S. Typhi, размером 2,3 т.п.н., в опытах in vitro установлено, что модуль отвечает за инвазию сальмонелл в эпителиальные клетки кишечника человека. Другие острова патогенности не были идентифицированы как модули SPI, но они кодируют гены, ответственные за вирулентность бактерии. [5, 26, 27].
Регуляторная система PhoP/PhoQ регулирует изменения липополисахаридов самой бактерии, что повышает ее резистентность к меняющимся условиям внешней среды и антимикробным препаратам, а также ведет к затруднению распознавания липополисахарида иммунной системой.
Гены spv (spvR, spvA, spvB, spvC и spvD) у S.typhimurium и S.enteritidis локализуются в крупной (размером 50-100 тысяч пар оснований) плазмиде, а у остальных сероваров в хромосоме. Кодируемые ими факторы обеспечивают распространение сальмонелл по организму, репродукцию в моноцитах, а также индукцию апоптоза последних.
Ген shdA размером в 6105 пар оснований проявляет гомологию с участками ДНК шигелл и диареегенных штаммов E.coli. Он обеспечил S. enterica адаптацию к теплокровным животным и регулирование интенсивности выделения бактерии с фекалиями.
Уникальный ген sifA состоит из 300 пар оснований и имеет более низкое суммарное содержание гуанина и цитозина (41%), чем другие части ДНК. Отвечает за образование филаментов, связывающих агента с мембраной фагосомы клеток эукариотов [4].
Итак, патогенные свойства микроорганизмов детерминированы в геноме. Причем некоторые из них имеют одну группу детерминант, другие - четыре, чем и определяются различия в патогенности сальмонелл [2].
Рецензенты:
Что такое сальмонеллез? Причины возникновения, диагностику и методы лечения разберем в статье доктора Александрова Павла Андреевича, инфекциониста со стажем в 14 лет.
Над статьей доктора Александрова Павла Андреевича работали литературный редактор Маргарита Тихонова , научный редактор Сергей Федосов и шеф-редактор Лада Родчанина

Определение болезни. Причины заболевания
Сальмонеллёз — это острое инфекционное заболевание желудочно-кишечного тракта с возможностью дальнейшей генерализации процесса (распространением заболевания по всему организму). Причина развития сальмонеллёза — различные серотипы бактерий рода Salmonella. К клиническим характеристикам сальмонеллёза относят синдром общей инфекционной интоксикации, синдром поражения желудочно-кишечного тракта (гастрит, энтерит), синдром обезвоживания, гепатолиенальный синдром (увелечение печени и/или селезёнки) и иногда синдром экзантемы (высыпания).
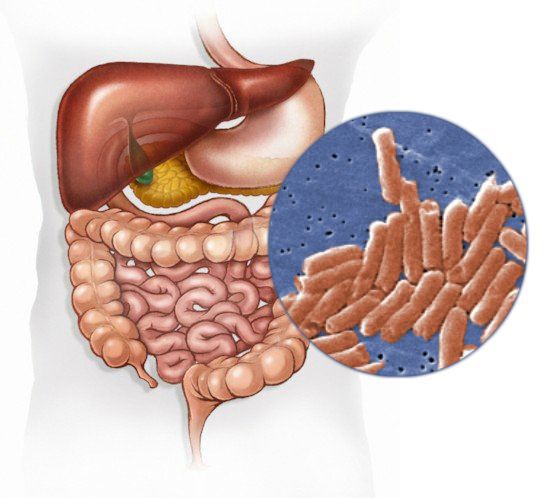
Возбудитель
семейство — кишечные бактерии (Enterobacteriaceae)
род — Сальмонелла (Salmonella)
Существует 7 подвидов (более 2500 сероваров). Наиболее актуальные серовары: typhimurium, enteritidis, panama, london.
Представлены следующей антигенной структурой:
- О-антиген (соматический, термостабильный);
- H-антиген (жгутиковый, термолабильный);
- К-антиген (поверхностный, капсульный);
- Vi-антиген (антиген вирулентности — степень способности штамма вызвать заболевание; является компонентом О антигена);
- М-антиген (слизистый).
К факторам патогенности (механизмам приспособления бактерий) относятся:
- холероподобный энтротоксин — интенсивная секреция жидкости в просвет кишки;
- эндотоксин (липополисахарид) — общее проявление интоксикации;
- инвазия — заражение.
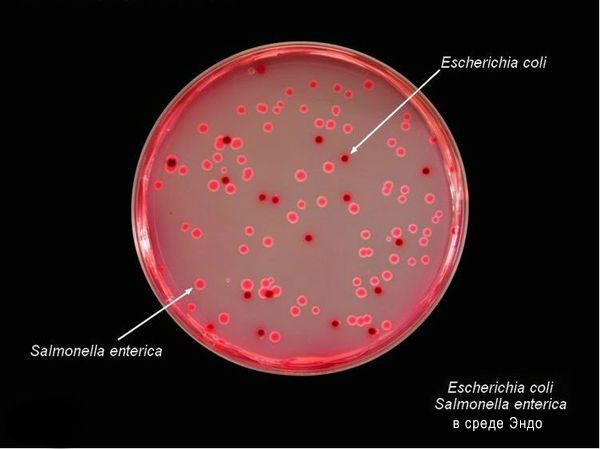
Тинкториальные свойства: разлагают глюкозу и маннит, образовывая кислоту и газ, продуцируют сероводород. Грамм-отрицательные палочки подвижны, спор и капсул не образуют. Растут на обычных питательных средах, образуя прозрачные колонии, на мясо-пептонном агаре с образованием колоний голубоватого цвета, на среде Эндо образуют прозрачные розовые колонии, на среде Плоскирева — бесцветные мутные, на висмут-сульфитном агаре — чёрные с металлическим блеском.
Высокоустойчивы во внешней среде (без агрессивных воздействий), активно размножаются в мясе и молоке (до 20 суток), в воде сохраняют жизнесособность до 5 мес., в почве — до 9 мес., в комнатной пыли — до 6 мес., в колбасе — до 1 мес., в яйцах — до 3 мес., в фекалиях сохраняются до 4 лет. При 56 °C погибают через 3 минуты, при кипячении мгновенно. Сальмонеллы, которые находятся в куске мяса массой 400 гр и толщиной до 9 см, погибают при его варке за 3,5 часа. Соление и копчение оставляет сальмонелл в живых. Воздействие кислот и хлорсодержащих дезинфицирующих средств вызывает их гибель. В последнее десятилетие появились штаммы сальмонелл, устойчивые ко многим антимикробным препаратам. [2] [5]
Эпидемиология
Зооантропоноз, распространённый повсеместно.
Источники инфекции: домашние животные (сами не болеют), птицы, человек (больной и носитель).
Резервуары инфекции и причина эпидемических вспышек сальмонеллеза: грызуны, дикие птицы, тараканы, улитки, лягушки, змеи.
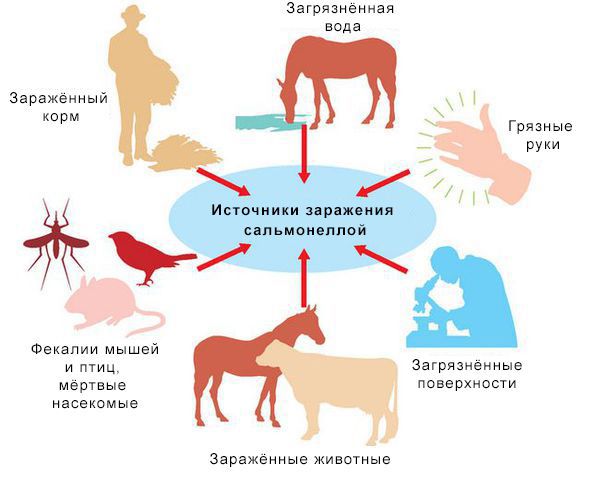
Механизм передачи: фекально-оральный (пути — алиментарный, т. е. через органы ЖКТ, водный, контактно-бытовой). В основном источниками заражения являются птицы, яйца и молочные продукты. Инфицирующая доза 10*5-10*8 микробных тел.
Факторы риска
- детский возраст до 5 лет;
- возраст до 12 месяцев, особенно высока вероятность заболеть без грудного вскармливания;
- иммунодефицит (в основном у младенцев и лиц старше 65 лет, а так же у пациентов с ВИЧ в стадии СПИДа, принимающих иммунодепрессивные препараты);
- регулярный приём препаратов, снижающих кислотность желудка;
- употребление сырого и недостаточно термически обработанного мяса, молочных продуктов и яиц;
- частый контакт с животными с несоблюдением правил гигиены;
- посещение стран с низким уровнем жизни.
В России в 2016 г. заболеваемость была – 26 на 100 тыс. населения, у детей в до 14 лет – 71 на 100 тыс. Для сравнения в США среднегодовая заболеваемость — 15 на 100 тыс. (1,35 миллиона заболеваний, 26 500 госпитализаций и 420 смертей ежегодно). Иммунитет строго типоспецифичен (возможно многократное инфицирование различными штаммами) и непродолжителен [2] [6] [9] [10] .
При обнаружении схожих симптомов проконсультируйтесь у врача. Не занимайтесь самолечением - это опасно для вашего здоровья!
Симптомы сальмонеллеза
Инкубационный период — от 6 часов (при алиментарном заражении) до 3 суток. При внутрибрюшном заражении (искусственно) — до 8 дней.
Начало заболевания острое (т. е. развитие основных синдромов происходит в первые сутки заболевания).
Salmonella is an important foodborne pathogen causing major public health problems throughout the world due to the consumption of contaminated food. Our previous studies have shown that deletion of glucose-inhibited division (gidA) gene significantly altered Salmonella virulence in both in vitro and in vivo models of infection. In Escherichia coli, GidA and MnmE have been shown to modify several bacterial factors by a post-transcriptional mechanism to modify tRNA. Therefore, we hypothesize that GidA and MnmE complex together to modulate virulence genes in Salmonella using a similar mechanism. To test our hypothesis, and to examine the relative contribution of GidA and MnmE in modulation of Salmonella virulence, we constructed gidA and mnmE single mutants as well as a gidA mnmE double mutant strain of Salmonella. Results from the in vitro data displayed a reduction in growth, motility, intracellular replication, and invasion of T84 intestinal epithelial cells in the mutant strains compared to the wild-type Salmonella strain. The in vivo data showed a significant attenuation of the mutant strains as indicated by the induction of inflammatory cytokines and chemokines, as well as in the severity of histopathological lesions in the liver and spleen, compared to mice infected with the wild-type strain. Also, a significant increase in the LD50 was observed in mice infected with the mutant strains, and mice immunized with the mutants were protected against a lethal dose of wild-type Salmonella. A pull-down assay indicated that Salmonella GidA and MnmE bind together, and HPLC analysis revealed that deletion of gidA and/or mnmE altered Salmonella tRNA modification. Overall, the data suggest MnmE and GidA bind together and use a post-transcriptional mechanism to modify tRNA to regulate Salmonella pathogenesis.
Copyright © 2013 Elsevier Ltd. All rights reserved.
Similar articles
Shippy DC, Eakley NM, Bochsler PN, Chopra AK, Fadl AA. Shippy DC, et al. Microb Pathog. 2011 Jun;50(6):303-13. doi: 10.1016/j.micpath.2011.02.004. Epub 2011 Feb 12. Microb Pathog. 2011. PMID: 21320585
Shippy DC, Fadl AA. Shippy DC, et al. BMC Microbiol. 2012 Nov 30;12:286. doi: 10.1186/1471-2180-12-286. BMC Microbiol. 2012. PMID: 23194372 Free PMC article.
Yim L, Moukadiri I, Björk GR, Armengod ME. Yim L, et al. Nucleic Acids Res. 2006;34(20):5892-905. doi: 10.1093/nar/gkl752. Epub 2006 Oct 24. Nucleic Acids Res. 2006. PMID: 17062623 Free PMC article.
Shippy DC, Fadl AA. Shippy DC, et al. Int J Mol Sci. 2014 Oct 10;15(10):18267-80. doi: 10.3390/ijms151018267. Int J Mol Sci. 2014. PMID: 25310651 Free PMC article. Review.
Meyer S, Wittinghofer A, Versées W. Meyer S, et al. J Mol Biol. 2009 Oct 2;392(4):910-22. doi: 10.1016/j.jmb.2009.07.004. Epub 2009 Jul 8. J Mol Biol. 2009. PMID: 19591841
Cited by 12 articles
Kouvela A, Zaravinos A, Stamatopoulou V. Kouvela A, et al. Int J Mol Sci. 2021 Aug 5;22(16):8409. doi: 10.3390/ijms22168409. Int J Mol Sci. 2021. PMID: 34445114 Free PMC article. Review.
Antoine L, Bahena-Ceron R, Devi Bunwaree H, Gobry M, Loegler V, Romby P, Marzi S. Antoine L, et al. Genes (Basel). 2021 Jul 24;12(8):1125. doi: 10.3390/genes12081125. Genes (Basel). 2021. PMID: 34440299 Free PMC article. Review.
Edwards AM, Addo MA, Dos Santos PC. Edwards AM, et al. Genes (Basel). 2020 Aug 7;11(8):907. doi: 10.3390/genes11080907. Genes (Basel). 2020. PMID: 32784710 Free PMC article. Review.
de Crécy-Lagard V, Jaroch M. de Crécy-Lagard V, et al. Trends Microbiol. 2021 Jan;29(1):41-53. doi: 10.1016/j.tim.2020.06.010. Epub 2020 Jul 25. Trends Microbiol. 2021. PMID: 32718697 Free PMC article. Review.
Что такое сальмонеллез? Причины возникновения, диагностику и методы лечения разберем в статье доктора Александрова Павла Андреевича, инфекциониста со стажем в 14 лет.
Над статьей доктора Александрова Павла Андреевича работали литературный редактор Маргарита Тихонова , научный редактор Сергей Федосов и шеф-редактор Лада Родчанина

Определение болезни. Причины заболевания
Сальмонеллёз — это острое инфекционное заболевание желудочно-кишечного тракта с возможностью дальнейшей генерализации процесса (распространением заболевания по всему организму). Причина развития сальмонеллёза — различные серотипы бактерий рода Salmonella. К клиническим характеристикам сальмонеллёза относят синдром общей инфекционной интоксикации, синдром поражения желудочно-кишечного тракта (гастрит, энтерит), синдром обезвоживания, гепатолиенальный синдром (увелечение печени и/или селезёнки) и иногда синдром экзантемы (высыпания).

Возбудитель
семейство — кишечные бактерии (Enterobacteriaceae)
род — Сальмонелла (Salmonella)
Существует 7 подвидов (более 2500 сероваров). Наиболее актуальные серовары: typhimurium, enteritidis, panama, london.
Представлены следующей антигенной структурой:
- О-антиген (соматический, термостабильный);
- H-антиген (жгутиковый, термолабильный);
- К-антиген (поверхностный, капсульный);
- Vi-антиген (антиген вирулентности — степень способности штамма вызвать заболевание; является компонентом О антигена);
- М-антиген (слизистый).
К факторам патогенности (механизмам приспособления бактерий) относятся:
- холероподобный энтротоксин — интенсивная секреция жидкости в просвет кишки;
- эндотоксин (липополисахарид) — общее проявление интоксикации;
- инвазия — заражение.
Тинкториальные свойства: разлагают глюкозу и маннит, образовывая кислоту и газ, продуцируют сероводород. Грамм-отрицательные палочки подвижны, спор и капсул не образуют. Растут на обычных питательных средах, образуя прозрачные колонии, на мясо-пептонном агаре с образованием колоний голубоватого цвета, на среде Эндо образуют прозрачные розовые колонии, на среде Плоскирева — бесцветные мутные, на висмут-сульфитном агаре — чёрные с металлическим блеском.

Высокоустойчивы во внешней среде (без агрессивных воздействий), активно размножаются в мясе и молоке (до 20 суток), в воде сохраняют жизнесособность до 5 мес., в почве — до 9 мес., в комнатной пыли — до 6 мес., в колбасе — до 1 мес., в яйцах — до 3 мес., в фекалиях сохраняются до 4 лет. При 56 °C погибают через 3 минуты, при кипячении мгновенно. Сальмонеллы, которые находятся в куске мяса массой 400 гр и толщиной до 9 см, погибают при его варке за 3,5 часа. Соление и копчение оставляет сальмонелл в живых. Воздействие кислот и хлорсодержащих дезинфицирующих средств вызывает их гибель. В последнее десятилетие появились штаммы сальмонелл, устойчивые ко многим антимикробным препаратам. [2] [5]
Эпидемиология
Зооантропоноз, распространённый повсеместно.
Источники инфекции: домашние животные (сами не болеют), птицы, человек (больной и носитель).
Резервуары инфекции и причина эпидемических вспышек сальмонеллеза: грызуны, дикие птицы, тараканы, улитки, лягушки, змеи.

Механизм передачи: фекально-оральный (пути — алиментарный, т. е. через органы ЖКТ, водный, контактно-бытовой). В основном источниками заражения являются птицы, яйца и молочные продукты. Инфицирующая доза 10*5-10*8 микробных тел.
Факторы риска
- детский возраст до 5 лет;
- возраст до 12 месяцев, особенно высока вероятность заболеть без грудного вскармливания;
- иммунодефицит (в основном у младенцев и лиц старше 65 лет, а так же у пациентов с ВИЧ в стадии СПИДа, принимающих иммунодепрессивные препараты);
- регулярный приём препаратов, снижающих кислотность желудка;
- употребление сырого и недостаточно термически обработанного мяса, молочных продуктов и яиц;
- частый контакт с животными с несоблюдением правил гигиены;
- посещение стран с низким уровнем жизни.
В России в 2016 г. заболеваемость была – 26 на 100 тыс. населения, у детей в до 14 лет – 71 на 100 тыс. Для сравнения в США среднегодовая заболеваемость — 15 на 100 тыс. (1,35 миллиона заболеваний, 26 500 госпитализаций и 420 смертей ежегодно). Иммунитет строго типоспецифичен (возможно многократное инфицирование различными штаммами) и непродолжителен [2] [6] [9] [10] .
При обнаружении схожих симптомов проконсультируйтесь у врача. Не занимайтесь самолечением - это опасно для вашего здоровья!
Симптомы сальмонеллеза
Инкубационный период — от 6 часов (при алиментарном заражении) до 3 суток. При внутрибрюшном заражении (искусственно) — до 8 дней.
Начало заболевания острое (т. е. развитие основных синдромов происходит в первые сутки заболевания).
Читайте также:

